表观遗传信息尤其是异染色质相关修饰能在细胞代间稳定传递,以此维持细胞命运的稳定性。这种现象被称为“表观遗传记忆”。在哺乳动物配子向早期胚胎转换的过程中,细胞命运发生剧烈重置,同时伴随着大规模的表观遗传重编程。尽管相关重编程图谱已被逐步绘制,但重编程过程中关键调控因子的鉴定及其调控机制仍是领域内亟待解决的核心问题。
2025年10月20日,清华大学生命学院颉伟研究组在《细胞-干细胞》(Cell Stem Cell)杂志上发表了题为“EZHIP限制PRC2非经典结合并调控H3K27me3代间继承和重编程”( EZHIP restricts noncanonical PRC2 binding and regulates H3K27me3 intergenerational inheritance and reprogramming)的研究论文。该研究在小鼠早期胚胎中鉴定出EZHIP(EZH inhibitory protein)是一个关键的表观遗传重编程因子,它控制了代间H3K27me3修饰的多层次重编程,包括全基因组范围内该修饰的擦除与重建,以及非经典印记表观记忆的传递。
H3K27me3是一种重要的抑制性组蛋白修饰,由PRC2(多梳复合物2)催化。在小鼠代间重编程中,H3K27me3呈现出高度动态变化:在卵母细胞中,H3K27me3分布于经典的发育基因启动子区域(称为多梳靶基因区域),同时也出现在基因间区,占据经常超过上百kb的区域(称为非经典H3K27me3结构域)。受精后,经典的H3K27me3在发育基因启动子处被快速擦除,直至着床前后才发生重建。然而非经典H3K27me3结构域在受精后仍能保留,并在着床前发育过程中短暂维持1。有趣的是,因为非经典H3K27me3结构域只存在于卵子来源的母源等位基因上,早期胚胎形成了由H3K27me3(而非DNA甲基化)控制的基因印记现象,被称为非经典基因印记2。为什么H3K27me3在代间重编程过程中在一些区域发生重置,同时在另一些区域发生遗传?这其中的调控因子是什么?这些都是未解之谜。
为解答这些问题,研究团队首先鉴定了卵母细胞与早期胚胎发育过程中PRC2的染色质结合区域。作者发现,在生长阶段的卵母细胞中,PRC2同时结合在多梳靶基因区域和非经典H3K27me3区域,活跃地催化H3K27me3修饰。然而在完全生长期的卵母细胞(FGO)中PRC2与染色质发生解离。受精后,PRC2在一细胞晚期重新出现在染色质上,并结合在非经典H3K27me3结构域。直至囊胚时期,PRC2才重新定位至多梳靶基因区域,启动H3K27me3的重建。
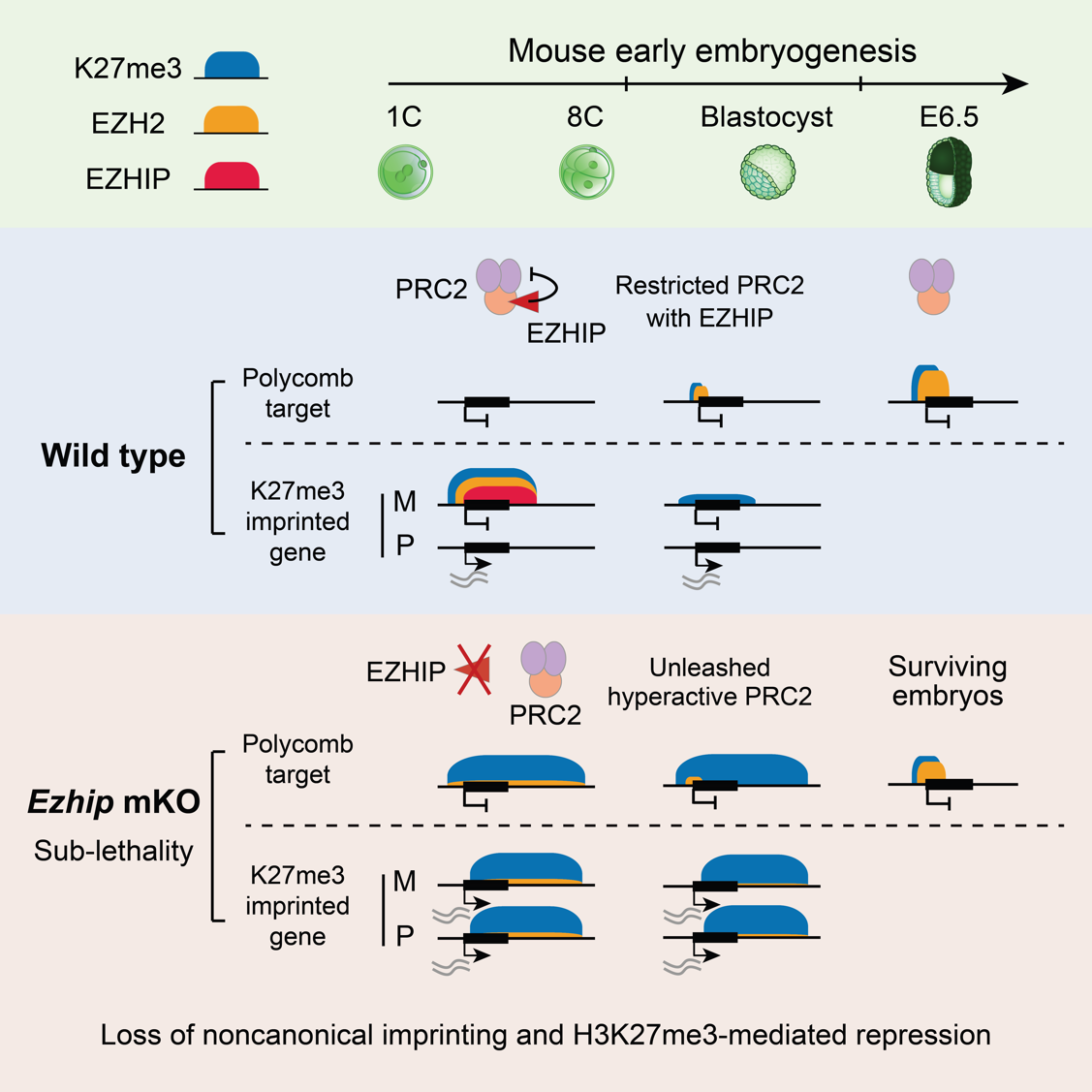
图. EZHIP调控PRC2非经典染色质结合和H3K27me3代间重编程
研究团队进一步证明,EZHIP在早期胚胎中调控PRC2非经典染色质结合与H3K27me3代间重编程。EZHIP是一种内源性PRC2抑制因子,仅在生殖细胞中特异性表达,而在其他细胞类型中被DNA甲基化沉默3。有趣的是,作者发现Ezhip mRNA在卵母细胞中大量积累,但处于翻译抑制状态。Ezhip仅在受精前后两天被活跃翻译,从而在着床前胚胎中表达丰度极高的蛋白。在野生型小鼠胚胎中,H3K27me3在着床前发生了全局性的去甲基化,而EZHIP缺失的小鼠胚胎中,去甲基化过程受阻,H3K27me3水平快速升高。更有趣的是,野生型胚胎中亲本来源的H3K27me3不对称性能维持到囊胚阶段,并介导非经典基因印记记忆;而EZHIP缺失后,H3K27me3不对称性在二细胞阶段已经完全丧失,导致非经典基因印记随之丢失。进一步的机制研究表明,着床前胚胎中EZHIP与PRC2能够共同结合在非经典H3K27me3结构域,在染色质上形成H3K27me3-PRC2-EZHIP复合体,以此限制PRC2的酶活和扩散4。EZHIP缺失后,PRC2不再被限制,继而在全基因组大部分区域催化了H3K27me3修饰,掩盖了几乎所有亲本来源的H3K27me3记忆。与常规认知相悖的是,这种广泛分布的H3K27me3修饰并未引起全局性基因沉默,反而让H3K27me3丧失了原本的抑制功能,导致非经典印记基因的异常激活和印记X染色体失活的失调。这可能是因为H3K27me3的效应因子(例如PRC1和PRC2)被大量出现的H3K27me3稀释,无法有效富集在正确的靶基因区域,从而导致基因抑制功能失效。另外,EZHIP缺失所造成的H3K27me3在全基因组范围内的过度积累还阻碍了后续多梳靶基因区域H3K27me3的重建,并伴随着胚胎植入后部分致死现象。
综上,本研究揭示了EZHIP是早期胚胎中关键的表观遗传重编程因子,调控了H3K27me3代间重编程的擦除、遗传和重建。同时,这些发现揭示了表观遗传记忆传递的基本原则,即异染色质相关酶活过低或者过高,均会导致表观遗传记忆和基因抑制功能的丧失(“过犹不及”)。
清华大学生命科学学院颉伟教授为本文通讯作者,清华大学生命科学学院2019级博士生曾埶天、2022级博士生孔凤为论文共同第一作者。清华大学生命科学学院2022级本科生徐子寒、博士后卢绪坤、2021级博士生李倩、博士后刘伯峰、实习生刘恕、颉伟实验室助理董丽君、刘玲、王文英也在该课题中作出了重要贡献。中国科学院生物物理研究所朱冰研究员为本课题提供了重要指导。该课题得到了清华大学实验动物中心的大力协助和支持。研究得到国家自然科学基金会、国家科技部重点研发计划、清华-北大生命科学联合中心和中国博士后科学基金的经费支持。颉伟教授是HHMI国际研究学者和新基石研究员。
参考文献
1. Zheng, H., Huang, B., Zhang, B., Xiang, Y., Du, Z., Xu, Q., Li, Y., Wang, Q., Ma, J., and Peng, X. (2016). Resetting epigenetic memory by reprogramming of histone modifications in mammals. Molecular cell 63, 1066–1079.
2. Inoue, A., Jiang, L., Lu, F., Suzuki, T., and Zhang, Y. (2017). Maternal H3K27me3 controls DNA methylation-independent imprinting. Nature 547, 419–424.
3. Piunti, A., and Shilatifard, A. (2021). The roles of Polycomb repressive complexes in mammalian development and cancer. Nat Rev Mol Cell Biol 22, 326–345. 10.1038/s41580-021-00341-1.
4. Jain, S.U., Rashoff, A.Q., Krabbenhoft, S.D., Hoelper, D., Do, T.J., Gibson, T.J., Lundgren, S.M., Bondra, E.R., Deshmukh, S., and Harutyunyan, A.S. (2020). H3 K27M and EZHIP impede H3K27-methylation spreading by inhibiting allosterically stimulated PRC2. Molecular Cell 80, 726–735. e727.
原文链接
https://www.cell.com/cell-stem-cell/fulltext/S1934-5909(25)00340-6