在过去几十年中,DNA 纳米技术已成为一个强大的平台,可以用于制造多样的纳米结构,从而在生命科学和材料科学领域开辟了众多的研究机会。其中 DNA 折纸方法已成为 DNA 纳米技术的主要突破之一。DNA 折纸结构的复杂性和可控性不断提高,展示了强大的自组装能力,可用于自下而上的纳米构建。尽管如此,快速生产高度定制的单链 DNA骨架链仍然是一个挑战。目前文献中提到的大多数 DNA 折纸结构都是使用具有固定长度和序列的 M13 噬菌体基因组 DNA 作为骨架链来构建的。
直接在折纸骨架链上自定义局部和全局序列特征无疑将带来更高的设计自由度和可控性。研究者们已经开发了多种技术来从双链 DNA模板生成单链骨架,包括不对称扩增、滚环扩增、酶切、微珠分离、丙烯酰胺标记分离和噬菌体介导的扩增等。但是,由于生产成本高和制备程序繁琐,自定义骨架链的广泛使用仍然受限。如果能开发一种简单而高效的生产流程,自定义双链模板和相应的单链骨架将更容易被更广泛的用户群体获取。
本研究提出了简便的生产流程来生成自定义长度和序列的单链骨架,并探索了多种新的折纸设计的可能性(图1)。这一流程的开发重点是使单链骨架生产程序标准化且简单直接。通过结合两种成熟的技术,即聚合酶循环组装(PCA)和λ外切酶消化,作者成功地生产了多种自定义的单链骨架。这两种技术的简单实施和可靠结果使这一骨架链生产流程既节省时间又节省成本。研究中展示的整个过程,包括 PCA 寡核苷酸序列从头设计、寡核苷酸合成、PCA组装和扩增反应、酶消化和纯化,总共需要不到7个工作小时。此外,PCA 寡核苷酸库可以重复使用,用以产生许多子单链骨架链变体,这显著降低了每个单链骨架的成本。
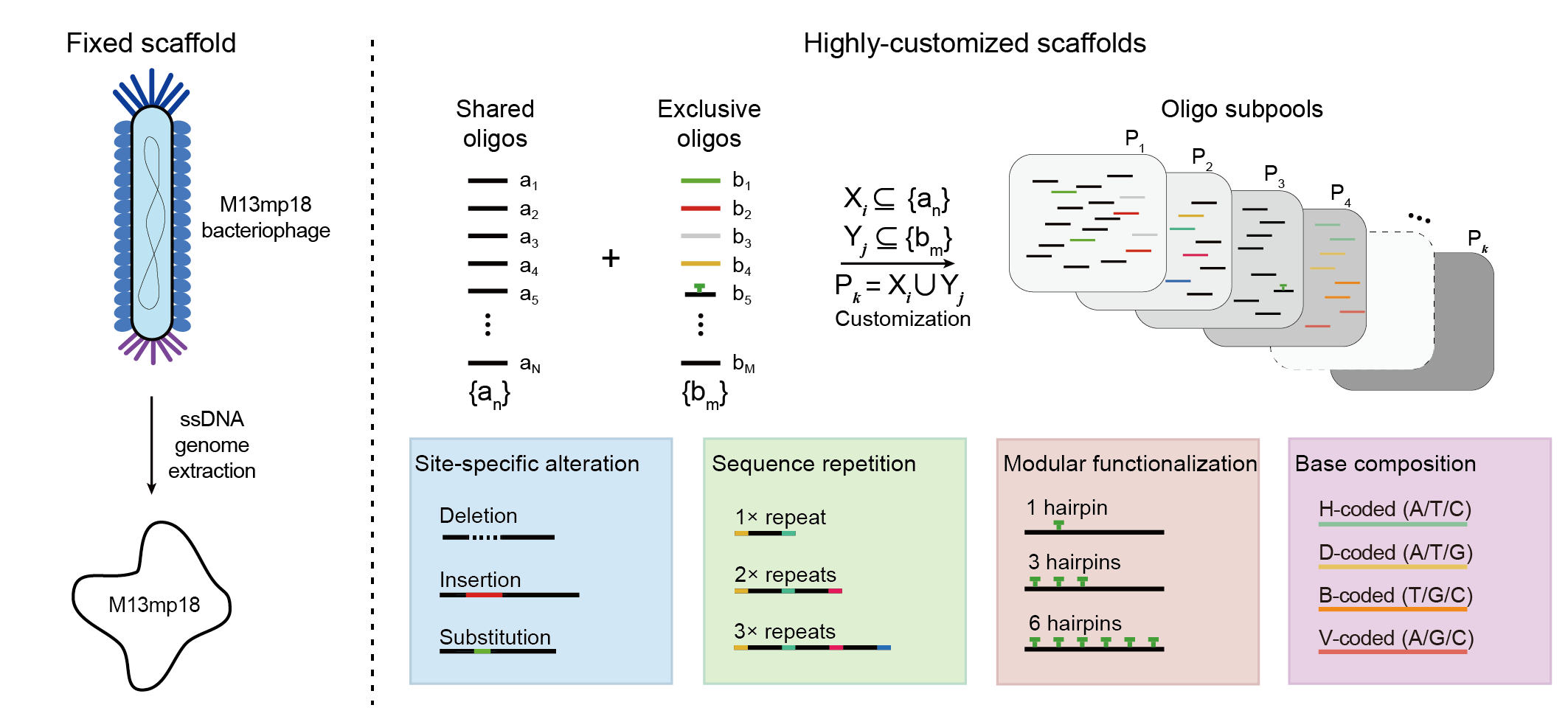
图 1. 来自 M13 噬菌体的固定序列骨架链和从头合成的自定义骨架链。在自定义骨架链中嵌入序列特征,将设计自由度扩展到了新的维度。
本研究的作者研究了多种嵌入自定义骨架链中的局部和全局序列特征的新的折纸设计和构建可能性。比如通过插入、删除和替换等方式灵活改变 DNA 骨架序列,构建不同形态的折纸结构;通过PCA寡核苷酸池的模块化排列,在骨架链上引入可自定义排列的功能序列,提高了折纸结构的功能化能力和序列排列的灵活性。
更重要的是,研究中测试了全局三字母编码骨架链的折叠行为,并在室温下展示了快速的折纸折叠结果(图2)。与其他研究在室温组装时需要甲酰胺和氯化钠的帮助不同,这一折纸结构室温折叠的动力学优势完全来自于三字母编码的序列,且折叠可以在室温下快速完成。研究中的实验也表明,不同的序列编码会导致完全不同的组装结果,应仔细考虑在全局和局部水平上的序列设计,以优化折纸折叠过程。
这项研究不仅证明了从头合成 DNA单链骨架生产流程的高效性和经济性,还展示了其在 DNA 折纸结构设计上的广泛潜力。通过灵活组合局部和全局序列特征的设计策略,作者成功拓展了 DNA 折纸的设计自由度。这将进一步释放 DNA 纳米结构的力量,从而扩展下游应用。同时,优化的PCA方案使得双链DNA片段从头合成得以高效和可靠的产生,其在合成生物学中也有一定应用前景。
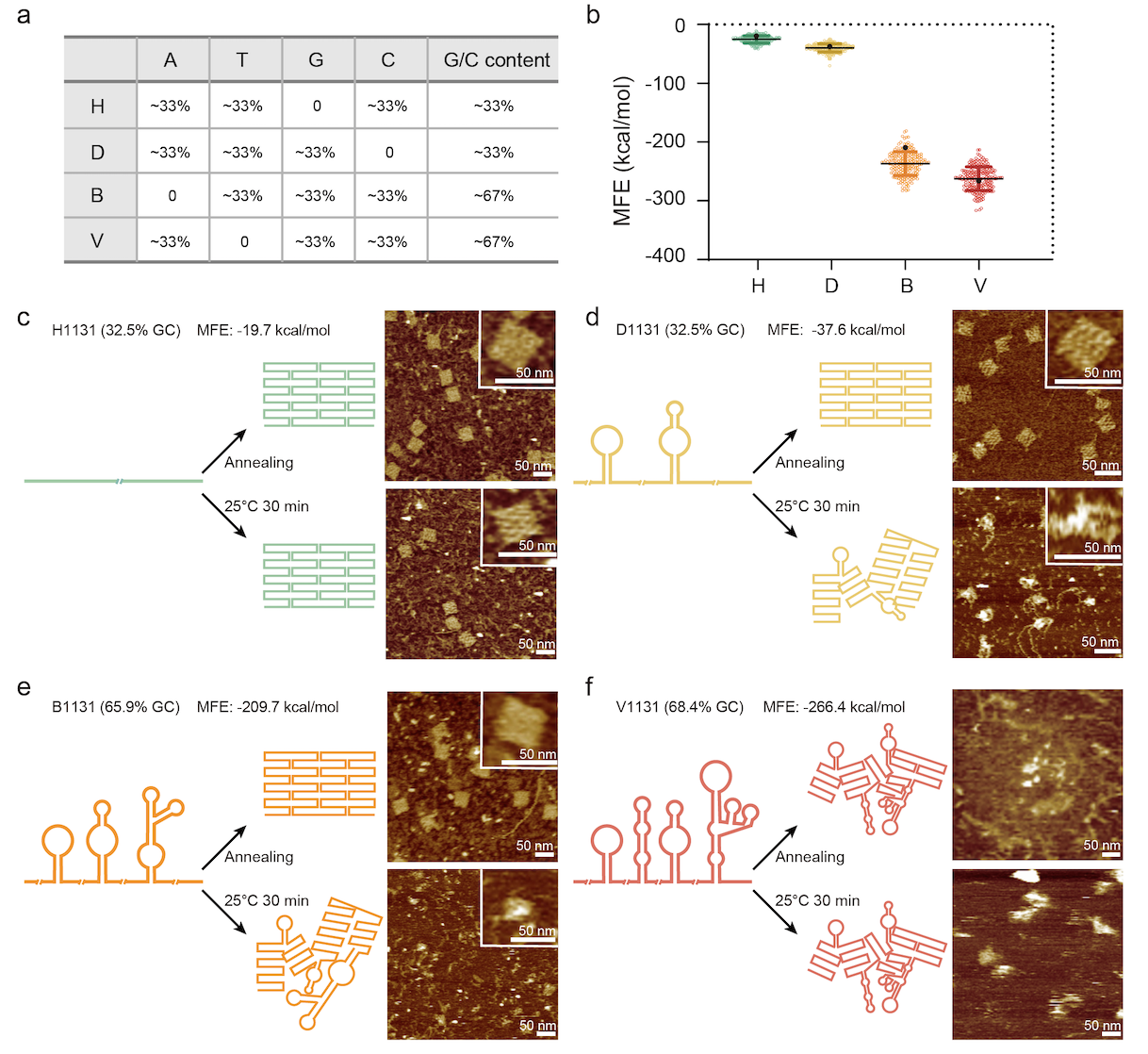
图2. 三字母编码的ssDNA骨架链。 (a) 不同编码方案的全局序列特征。 (b) 计算机模拟生成的不同编码方案骨架链的最小自由能(MFE)。最高的 MFE 显示为 H 编码的骨架链(绿色),中等 MFE 显示为 D 编码(黄色)和 B 编码(橙色)的骨架链,最低的 MFE 显示为 V 编码的骨架链(红色)。 (c) H 编码骨架链的折纸折叠结果。 (d) D 编码骨架链的折纸折叠结果。 (e) B 编码骨架链的折纸折叠结果。 (f) V 编码骨架链的折纸折叠结果。左侧:折叠情况图示;右侧:原子力显微镜结果,展示了典型冷却程序(右上)和室温(右下)下的折纸折叠结果。
该研究成果由清华大学生命科学学院魏迪明分子设计课题组(MADlab)完成。论文题目为“利用从头合成的骨架链扩展 DNA 折纸设计自由” (Expanding DNA origami design freedom with de novo synthesized scaffolds),于2024年5月27日在线发表于《美国化学学会志》(Journal of the American Chemical Society)。
清华大学生命学院2018级博士生午红蕊和2017级博士毕业生张天晴为本文的共同第一作者。张天晴博士和清华大学生命学院魏迪明副教授为本文的共同通讯作者。该研究得到了科技部、清华大学春风基金、清华-北大生命科学联合中心等基金资助。
论文链接:
https://pubs.acs.org/doi/10.1021/jacs.4c03148