2019年3月13日,清华大学陈柱成课题组和北京大学高宁课题组合作在《自然结构与分子生物学》(Nat. Struct. Mol. Biol)杂志发表题为“ISWI-核小体复合物结构揭示一种保守的染色质重塑机理”(“Structures of the ISWI-nucleosome complex reveal a conserved mechanism of chromatin remodeling”)的研究论文。该工作采用冷冻电镜手段,解析了不同功能状态下染色质重塑蛋白ISWI与其底物核小体结合的高分辨结构,揭示了ISWI与Snf2保守的催化核小体滑移的机制,推翻了染色质重塑的组蛋白形变假说。
ISWI驱动核小体在基因组DNA上滑动以重塑染色质结构,参与调控基因转录、异染色质形成、X-染色体失活及其它重要的染色质活动。然而,此前没有高分辨的ISWI-核小体复合物的三维结构,人们对于ISWI的工作机理的尚不清楚。
陈柱成课题组在2016年分析了ISWI在没有结合核小体时的自抑制机理(Yan,Nature 2016)。在这基础上,本研究解析了ADP-BeFx和ADP状态下ISWI-核小体复合物的高分辨结构(图a-b)。结合核小体之后,ISWI的催化核心发生148°的旋转,解除ISWI蛋白的自抑制,这揭示了核小体激活ISWI的机理(图c)。通过对比ADP-BeFx和ADP状态下ISWI-核小体复合物的结构,他们发现ATP水解后核小体SHL2处DNA产生了1 bp隆起(核小体DNA的分辨率达到3.3 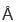 ),展示了染色质重塑过程中“DNA波”的具体存在形式(图d)。这与Snf2高度相似。
),展示了染色质重塑过程中“DNA波”的具体存在形式(图d)。这与Snf2高度相似。
有文献报道ISWI结合核小体之后诱使组蛋白核心发生了扭曲,这种形变为ISWI发挥染色质重塑功能所必需。组蛋白形变假说引起了领域内的广泛关注。然而,本研究并没有发现组蛋白形变(图e)。而且通过更温和、彻底的蛋白交联,作者发现ISWI发挥染色质重塑过程不依赖的组蛋白扭曲(图f)。这个工作与陈柱成实验室同期的有关Snf2的研究一起(Li, Nature 2019),提出了一个高度保守的染色质重塑普适机理。
清华大学陈柱成研究员和北京大学高宁教授为本文的共同通讯作者,清华大学生命科学学院博士生严丽娟和PTN博士生吴昊为论文的共同一作。该研究得到国家重大研发计划、国家自然科学基金委、北京市高精尖结构生物学中心和生命联合中心项目的经费支持,以及清华大学电镜平台和计算平台、北京大学电镜实验室(冷冻电镜平台)以及北京大学高性能计算中心的工作支持。
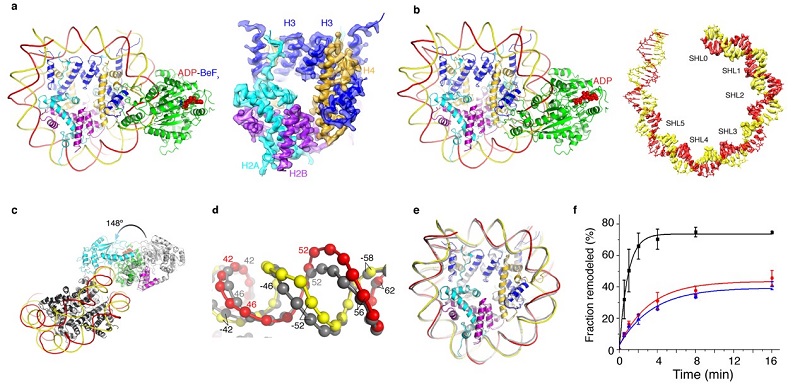
图 1 ISWI-核小体复合物在不同功能状态下的高分辨结构 (a) ADP-BeFx状态; (b) ADP 状态; (c) ISWI被核小体激活的机理;(d) ISWI(ADP)引起的DNA形变;(e) ISWI作用下,组蛋白核心保持正常结构;(f) 组蛋白形变不是ISWI介导的染色质重塑的必须条件。
论文链接: https://www.nature.com/articles/s41594-019-0199-9